![]()
4.3 細胞が目覚める分子メカニズムの追究
真核生物の細胞は、必ずしも常に増殖しているわけではありません。周囲の環境次第では活発に増殖することができますが、生存に適していない環境では目立った活動を停止して「休眠」状態になります。休眠は決して細胞の死ではなく、休眠した状態で長きにわたり生存を維持することができます。そして、周囲の環境が生存に適した状態になると、細胞は目覚めて「増殖」を開始します。例えば、植物の種子は休眠状態にあり、環境の変化に応じて発芽します。動物細胞でも休眠状態にある細胞が何らかの原因によって目覚めてしまうことが、がん化の1つの契機になると言われます。
このように、細胞の増殖と休眠は、周囲の環境に応答して自身の運命を決める細胞にとって極めて重要な生存戦略だといえます。にもかかわらず、どのような分子 (遺伝子) がその運命を選択しているのか、メカニズムについてはほとんどわかっていません。
そこで我々は、従来の遺伝学と新たに取り入れた先端計測の実験手法をもとに実験をおこない (wet解析) 、そこで得られたデータに対してバイオインフォマティクス解析を実施する (dry解析) ことで、細胞が休眠から目覚める分子機構のモデルを提案することに成功しました (Tsuyuzaki et al., Nature communications, 2020)。今後、この情報解析から導かれたモデルが正しいものかを検証するために、さらに遺伝学的・分子細胞生物学的な実験を展開していきます (wet解析) 。
このように、我々は伝統的・先端的な実験 (wet解析) と、統計学や人工知能AIを含めた情報科学 (dry解析) を相互的に組み合わせて止揚する弁証法的解析を反復することで、細胞の運命決定に秘められた謎を解明していきます。
4.3.1 細胞の休眠と目覚め
真核生物の細胞は、細胞分裂による「増殖」を繰り返します。しかし、細胞をとりまく周囲の環境が変化すると、それに応じて細胞周期から抜け出し、「休眠」状態に入り、それ以降は分裂しません。菌類、植物、動物にみられる真核細胞は常に増殖しているわけではなく、休眠状態にある細胞もしばしばみられます。そのような休眠細胞の中には、環境が変化すると「目覚め」て、増殖状態に入るものがあります。
例えば、休眠細胞の代表的なひとつの例は植物の種子であり、種子として休眠することで、低温や乾燥などの生存に不利な環境でも長期間生存できます。そして、細胞増殖に適した環境条件になると種子は目覚め (発芽し) 、植物体になります。また、私たちの体を構成する細胞の多くは休眠状態にあります。これらの一部が何らかの理由により目覚めることが細胞のがん化の一因だと考えられています。
このような細胞の目覚めはどのような分子メカニズムで起こっているのでしょうか。目覚めのメカニズムを解明できれば、基礎科学の追究として興味深いうえに、農学的·医学的な応用が可能になると考えられます。例えば、不適切な細胞の目覚めを抑制することができれば、がんの治療法のひとつになり得ます。ここで我々は、休眠と目覚め研究をおこなうためのモデル生物として、動物や植物よりも簡単に実験が可能であり、休眠と目覚めを人為的に簡単にコントロールできる分裂酵母(Schizosaccharomyces pombe)を選び、研究を展開しています。
4.3.2 分裂酵母の発芽
分裂酵母は、栄養がある環境では体細胞分裂を繰り返して増殖します (図4-4)。しかし、栄養が不足した状態になると増殖を停止して減数分裂を行い、配偶子である胞子を産生します。 胞子では細胞内の代謝が抑えられており、休眠することで不利な環境下でも生存を維持できます。しかし、栄養が与えられると、目覚めて体細胞分裂による増殖状態を開始します。この現象を胞子の「発芽」と呼びます。
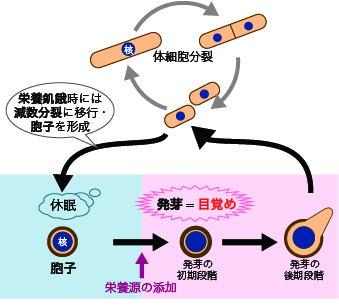
図4-4 分裂酵母の休眠と目覚め
酵母は、栄養のある環境では体細胞分裂によって増殖するが、栄養がなくなると増殖を停止し、減数分裂を経て胞子 (配偶子) を産生する。胞子は生育に不利な環境に耐えられる休眠細胞であるが、再び環境中に栄養が与えられると目覚めて (発芽して) 、体細胞分裂による増殖過程に入る。
我々は、目覚めの分子メカニズムを解明するためには、休眠から発芽に至る過程において、細胞内にどの遺伝子がどれくらい発現しているかを調べること、つまり各遺伝子のmRNA量を調べることがブレーク·スルーになると考えました。特に、発芽の最初期の段階に発現を増加 (または減少) させる遺伝子こそが、細胞の目覚めに重要な意味をもつ遺伝子だと想定しました。
細胞内において、それぞれの遺伝子がどれくらいの量で発現しているかを一覧にしたデータを遺伝子発現プロファイルと呼びます。遺伝子発現プロファイルを作成するためには、次世代シーケンサーを使用してmRNAの配列を大量に解読するRNA-seq (RNAシーケンシング) とよばれる方法が近年は一般的です。すなわち、発芽の初期段階にある胞子を多数準備して、その集団からmRNAを回収し、RNA-seqを行えば、発芽における遺伝子発現プロファイルを作成できるはずです。
しかし、ここで問題がありました。従来のRNA-seq実験には技術上の制約があり、多数の酵母細胞からmRNAを大量に調製する必要があったのです。そのために、発芽し始めた胞子を大量に集めようと思っても、発芽のタイミングには個体差があるため、胞子の集団に対して一斉に発芽を誘導することができず、その時期の胞子を大量に集めることができません。
そこで我々はシングルセルRNA-seq (scRNA-seq)という手法を分裂酵母研究に開発・導入することで、この問題を解決しました。近年開発が進んできたscRNA-seq法では、1細胞から単離した微量なmRNAをもとに細胞内mRNA量を調べることができるため、大量の胞子を準備する必要も、それらの発芽のタイミングを同調させる必要も一切ありません。我々が本研究に着手した段階では、分裂酵母を用いてscRNA-seqをおこなった報告は1例もなかったので、まずその技術を開発するところからおこないました。特に胞子におけるscRNA-seqは我々の論文 (Tsuyuzaki et al., Nature Communications, 2020) が世界で初の発表例となります。
4.3.3 バイオインフォマティクスを用いた解析
我々は、胞子を1細胞ごと選び取り、それぞれの細胞から個別にmRNAを採取してそのmRNA配列と存在量を一覧にしたscRNA-seqプロファイルを作成しました。64個の胞子を1個ずつ選び取ったので、64個のscRNA-seqプロファイルが作成できました。分裂酵母の細胞には、データベースに登録されている約7,000個の遺伝情報 (便宜的に「7,000遺伝子」と呼ぶことにします) が存在します。発芽時にそれらの遺伝子の各々がどれくらいの量で発現しているのか、64細胞×7,000遺伝子という配列情報のビッグデータを得ることができました。
さて、思い起こしてみると、我々は発芽過程の最初の時期にあたる細胞に注目して、その中でどの遺伝子の発現が変動するのかを見たかったわけです (→ 4.2.1、4.2.2 節)。では、顕微鏡下で選んだ64個の胞子のなかで、どの細胞が目覚めの一番初期にあたるのでしょうか。顕微鏡下で細胞を選ぶ際は、どの胞子も形態があまり違わないので、今回選んだ64個のなかでどの細胞が発芽の最初期にあたるものかをそこで判断することはできませんでした。そこで、バイオインフォマティクス(生物情報科学)の技法を駆使して、64個の細胞のなかでどれが発芽の初期で、どれが中期·後期であるのかを見分ける試みを導入しました。つまり、細胞の見た目では決して分からない「休眠からの目覚め具合」を、それらの遺伝子発現プロファイルの情報を人工知能(AI)解析することで計算上推定できるというアイデアです (図4-5)。
64個の発現プロファイルを計算により比較して、似ている順にそれらを並べることによって、「おそらく64個の細胞は時間とともにこのような順番で変化していったのだろう」という変化の流れを推測する仮想的な時間軸(バーチャル・タイムライン)を推定することに成功しました (Tsuyuzaki et al., Nature communications, 2020; Tsuyuzaki et al., Current Genetics, 2021) 。
このようにして発芽最初期の細胞がどれであるか分かったので、最初期に発現量が激しく変動する遺伝子をいくつか決定することができました。そのなかに、ヒストンH3をコードする遺伝子の一つであるhht1が含まれていることを突き止めました。
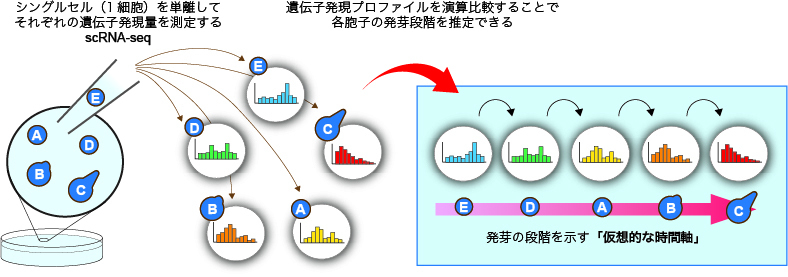
図4-5:バイオインフォマティクスを用いたバーチャル·タイムラインの推定
胞子が目覚めのどの段階にあるのか (目覚めの初期か中期か) は、顕微鏡下で細胞の形態をみても視覚的には判別できない。そこで、それぞれの細胞からscRNA-seqにより遺伝子発現量を測定し、バイオインフォマティクス解析をおこない、それぞれの細胞が目覚めの時系列のなかでどの段階にあるのかを示す、仮想的な時系列 (バーチャル・タイムライン) を推定することに成功した。これにより、選んだ64個の胞子の中で、目覚めの最初期にある細胞がどれであるかを探し当てることができるようになった。
4.3.4 目覚めを促進する遺伝子として浮かび上がったヒストンH3
ヒストンはDNAに結合するタンパク質であり、遺伝子の発現を調節する役割を担うといわれています。8個のヒストンタンパク質が会合してヒストン8量体を形成し、これにDNAが巻き付いた構造はヌクレオソームと呼ばれます (図4-6) 。染色体全域にわたり、DNAとヒストン8量体はこのようなヌクレオソーム構造を作っていますが、染色体 (クロマチン) におけるヒストン8量体の結合状態をコントロールすることで、遺伝子の発現量が変化するといわれます。
ヒストン8量体を構成する成分の一員としてヒストンH3が存在します。分裂酵母では3つのヒストンH3遺伝子 (hht1、hht2、hht3 ) がゲノム上に存在します。それら3種類の遺伝子が指定するタンパク質のアミノ酸配列は完全に同一であるため、これら3つの遺伝子が単に重複して存在するだけなのか、あるいは機能的に使い分けられているのかは知見がありませんでした。我々は今回の解析により、3つのヒストンH3遺伝子の中でhht1が発芽の初期段階に発現量が減少し、その後、発芽の中期~後期段階で再び上昇することを発見しました。これに対して、hht2とhht3は発芽の過程を通して発現量があまり変わりません。
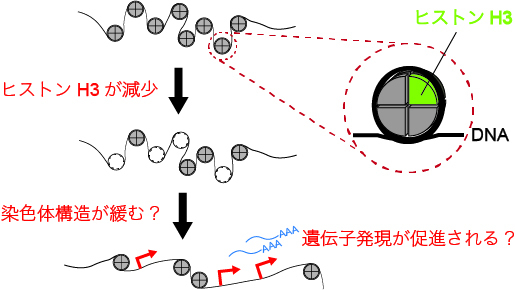
図4-6 ヌクレオソーム状態の変化による遺伝子発現の調節の可能性
4.3.5 ヒストンH3の発現は減るべきか、増えるべきか
次にhht1を遺伝子破壊 (ノックアウト) して、hht1が一切発現しない胞子を作製して観察したところ、体細胞分裂による増殖過程には異常がなかった反面、胞子からの発芽は顕著に遅れることが分かりました。つまり、ヒストンH3をコードする3つの遺伝子のうち、hht1は細胞が目覚めるために重要な遺伝子だといえます。3つのヒストンH3遺伝子は完全に同一なアミノ酸配列を指定するので機能が同じようにみえるものの、これらが働く時期·場面が異なるため、絶妙に使い分けされていることが我々の解析から明らかになってきました。
このように、発芽を促進するためにはhht1遺伝子が存在する(=発現する)ことが求められる一方で、4.3.4節で述べたように、発芽の最初期にはhht1の発現量がむしろ減少しています。hht1の発現量が増加するのではなく、減少することに何か意味があるのでしょうか? この問いに答えるために、人工的に遺伝子改変を施し、目覚め時にhht1の発現が減少しない細胞を作製しました。この細胞は、hht1が一切発現しないノックアウト細胞と同様に、発芽が著しく遅れる欠陥を示しました。これらの実験結果から、発芽の最初期にはhht1の発現量が減少すること、その後、発芽の中期~後期にはhht1の発現が上昇すること、そして効率的な発芽のためにはhht1発現の減少と上昇の両方が欠かせないことが示唆されました。では、ヒストンH3の発現量が低下することがなぜ細胞の目覚めに繋がるのでしょうか?
一般に、DNAに巻き付くヒストン8量体が減少してクロマチン構造が緩くなる (クロマチンがオープン状態になる) と遺伝子の発現が促進されて、逆に何らかの原因でクロマチン構造が強固になる (クローズ状態) と、遺伝子発現は抑制される傾向にあることが知られています。このことから類推すると、発芽の初期にヒストンH3の量が減少することでクロマチン構造が緩み、ゲノム全体で遺伝子発現が上昇することが想像されます (図4-6) 。このような大規模な遺伝子発現の誘導によって、休眠からの目覚めが進行すると考えられます。
4.3.6 これからの展望
現在は、これらのバイオインフォマティクス解析 (dry解析) から導き出された上記の推論が正しいのかを検証するために、クロマチン状態を可視化する実験 (wet解析) を計画しています。さらに、バイオインフォマティクス解析によって発芽初期に発現変動する遺伝子として見つけ出したhht1以外の遺伝子についても、実験による検証 (wet解析) を進めて、細胞がどのような制御を受けて目覚めていくのか、運命決定の全体像を描いていきたいと考えます。最初に述べたように、細胞の目覚めは酵母のみならず、動植物の細胞にも見られる普遍的な生命現象です。細胞の休眠と目覚めにおいては、環境への適応·細胞寿命·運命決定など、様々な細胞現象のクロストークが密接に起きていると思われ、研究の重要性は今後高まっていくと確信しています。
Related Publications:
Hayato Tsuyuzaki, Ryosuke Ujiie, Masamitsu Sato
Current Genetics (2021) 67(4):519-534. DOI:10.1007/s00294-021-01177-0
シングルセルRNA-seq解析から明らかになった分裂酵母の目覚め機構
露崎隼, 細川正人, 竹山春子, 佐藤政充
バイオサイエンスとインダストリー(B&I) (2020) 78(6):507-509.
Time-lapse single-cell transcriptomics reveals modulation of histone H3 for dormancy breaking in fission yeast
Hayato Tsuyuzaki, Masahito Hosokawa, Koji Arikawa, Takuya Yoda, Naoyuki Okada, Haruko Takeyama and Masamitsu Sato
Nature Communications (2020) 11:1265. DOI:10.1038/s41467-020-15060-y
